Genregulation als Ansatzpunkt für Krebstherapien
Neue Untersuchungsmethode zur Entschlüsselung komplexer epigenetischer Netzwerke
Für die Entstehung und Aufrechterhaltung der unkontrollierten Zellteilung in Tumoren ist häufig das aus dem Gleichgewicht geratene, komplexe Zusammenspiel der regulatorischen epigenetischen Netzwerke verantwortlich. Forschende am Institut für Biochemie und Technische Biochemie in Stuttgart haben ein neues Untersuchungssystem entwickelt, mit dem sich essenzielle Komponenten identifizieren lassen, die als Angriffspunkte für Krebsmedikamente dienen können.
Alle unsere Körperzellen enthalten die identische genetische Information, trotzdem besitzen wir mehr als 300 verschiedene Zelltypen mit unterschiedlichen Funktionen. Die erforderliche Differenzierung während der Entwicklung von der befruchteten Eizelle zum Organismus ist nur durch ein reguliertes Ablesen (Transkription) der Gene möglich. Dieser Prozess wird durch die Epigenetik gesteuert, die eine übergeordnete Regulationsebene bildet. Sie bestimmt, wann und in welchen Zellen einzelne Gene genutzt werden.
Träger der genetischen Information sind die 46 Chromosomen im Kern jeder Zelle. Sie bestehen aus einem langen Nukleinsäurestrang, der mithilfe von speziellen Proteinen, den Histonen, verpackt wird. „Die Epigenetik steuert den Verpackungsgrad der DNA“, erklärt Dr. Philipp Rathert, Gruppenleiter am Institut für Biochemie und Technische Biochemie (IBTB) der Universität Stuttgart. „Eng verpackte DNA kann nicht abgelesen werden, offene Regionen hingegen sind zugänglich.“ Der Zustand wird durch kleine chemische Veränderungen (Modifikationen), wie beispielsweise das Anhängen von Acetylresten (-C(O)CH₃) oder Methylgruppen (-CH3) an die Histone, beeinflusst. Diese Modifikationen erzeugen vor allem neue Bindungsstellen für regulatorische Proteine und können die Transkription sowohl fördern als auch hemmen. Die Methylierung von DNA-Bausteinen (vorrangig Cytosin) ist ein weiterer wichtiger Mechanismus, der in regulatorischen Bereichen von Genen (Promotoren, Enhancer) dazu führt, dass diese stumm geschaltet werden.
Komplexe epigenetische Netzwerke regulieren die Zellfunktion
 Dr. Philipp Rathert, PhD, erforscht die komplexen Zusammenhänge der Regulation des epigenetischen Enzyms LSD1. © P. Rathert
Dr. Philipp Rathert, PhD, erforscht die komplexen Zusammenhänge der Regulation des epigenetischen Enzyms LSD1. © P. RathertWährend der Entwicklung eines Organismus werden epigenetische Markierungen gesetzt und wieder rückgängig gemacht, je nachdem in welchem Stadium sich die Zellen befinden. Im ausdifferenzierten Zustand hängt die Zellfunktion dann vom ausgewogenen Zusammenspiel dieser Modifikationen ab und wird bei der Zellteilung an die Tochterzellen weitergegeben. „Wenn dieses dynamische System der Regulation aus dem Gleichgewicht gerät, können Zellen wieder de-differenzieren und beginnen, sich unkontrolliert zu teilen. Dies ist der erste Schritt zur Krebsentstehung“, beschreibt Rathert das Problem. Seine Arbeitsgruppe in der Abteilung Biochemie untersucht unter anderem die Lysin-spezifische Histon-Demethylase LSD1, die in einer Vielzahl an Krebsarten, wie beispielsweise der akuten myeloischen Leukämie (AML) sowie Brust-, Blasen- und Lungenkrebs, eine wichtige Rolle spielt. Das Enzym ist essenziell für die Embryonalentwicklung und leistet außerdem einen wichtigen Beitrag zur Aufrechterhaltung des epigenetischen Gleichgewichts in ausdifferenzierten Zellen.
LSD1 als Angriffspunkt bei Krebstherapien
LSD1 ist Bestandteil verschiedener Proteinkomplexe und entfernt Methylierungen an Lysin 4 (K4) des Histons H3. Abhängig von den Komplexpartnern kann dies Gene aktivieren oder abschalten, die genaue Regulation des Enzyms ist allerdings bisher noch weitgehend unklar. Da in vielen Krebsarten erhöhte Mengen an LSD1 auftreten, bzw. im Labor nachgewiesen wurde, dass LSD1-Inhibitoren das Krebswachstum reduzieren können, werden diese derzeit in Klinischen Studien untersucht. Vor allem bei AML und dem kleinzelligen Lungenkarzinom (Small-cell lung cancer SCLC) zeigen die Inhibitoren eine gute Wirksamkeit. „Monotherapien erzeugen allerdings langfristig häufig Resistenzen und haben aufgrund der recht hohen Dosis oft starke Nebenwirkungen. Deshalb geht man immer mehr zu Kombinationstherapien über, bei denen dann z. B. mehrere Partner eines Komplexes als Angriffspunkt dienen. Hier haben wir angesetzt und uns auf die Suche nach essenziellen Interaktionspartnern von LSD1 gemacht“, erläutert der Epigenetiker die Strategie seines Labors.
Neuartiges Untersuchungssystem zur Identifizierung von epigenetischen Co-Regulatoren
Um Licht in das komplexe regulatorische Netzwerk um LSD1 zu bringen, entwickelte der Wissenschaftler mit seinem Team im ersten Schritt ein fluoreszenzbasiertes Reportergen-System, mit dem die Aktivität des Enzyms in lebenden Zellen analysiert werden kann. Hierbei wird die genetische Information für ein rot fluoreszierendes Protein (mCherry) unter der Kontrolle eines synthetischen Promotors mit speziellen Tet-Repressor- (tetR) Bindesequenzen in Mauszelllinien eingebracht, sodass die Zellen intensiv rot leuchten. Des Weiteren produzieren die Zellen das humane LSD1-Protein, das mit dem reversen tetR-Bindeprotein verknüpft ist. Durch Zugabe von Doxycyclin wird die Bindung von LSD1 und damit auch seiner Komplexpartner an den Promotor von mCherry initiiert. Dies verändert die Methylierung der benachbarten Histone und hat zur Folge, dass die Produktion des fluoreszierenden Proteins unterdrückt wird.
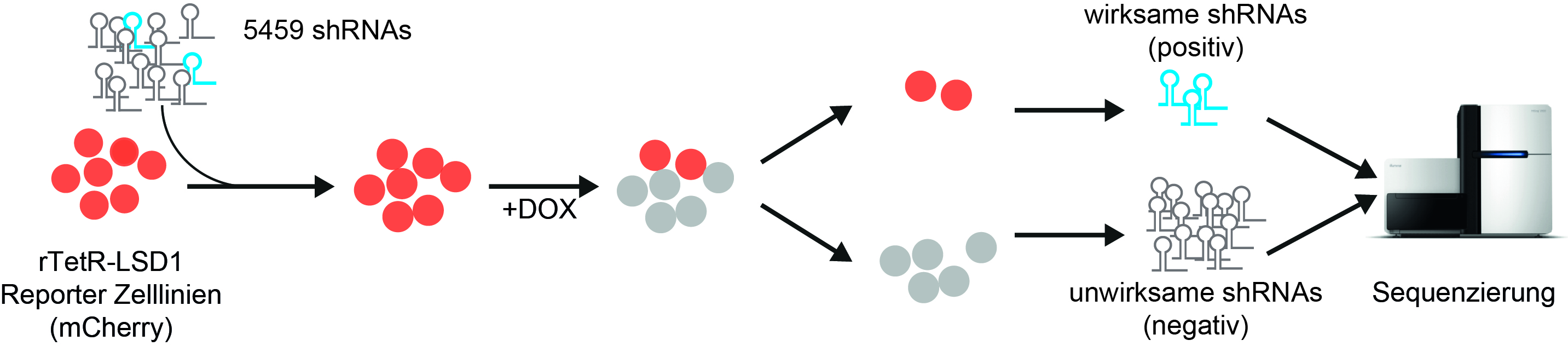 ChECS (Chromatin Effector Coregulator Screen) Methode: Rot fluoreszierende Reporter-Zelllinien, die LSD1 fusioniert mit dem reversen tetR-Bindeprotein produzieren, werden mit einer Sammlung aus shRNA-Molekülen transduziert. Nach Zugabe von Doxycylin (DOX) bindet LSD1 an den Promotor des Gens für mCherry und schaltet es aus (graue Zelllinien). Wird ein für die Funktion von LSD1 essenzielles Protein durch shRNAs ausgeschaltet, leuchten die Zellen weiterhin rot. Die entsprechende genetische Information kann mittels Sequenzanalyse identifiziert werden. Quelle: „Workflow describing the ChECS screening“, P. Rathert, https://doi.org/10.1093/nar/gkab180, Oxford University Press, CC-BY-NC 4.0, bearbeitet von R. Menßen-Franz.
ChECS (Chromatin Effector Coregulator Screen) Methode: Rot fluoreszierende Reporter-Zelllinien, die LSD1 fusioniert mit dem reversen tetR-Bindeprotein produzieren, werden mit einer Sammlung aus shRNA-Molekülen transduziert. Nach Zugabe von Doxycylin (DOX) bindet LSD1 an den Promotor des Gens für mCherry und schaltet es aus (graue Zelllinien). Wird ein für die Funktion von LSD1 essenzielles Protein durch shRNAs ausgeschaltet, leuchten die Zellen weiterhin rot. Die entsprechende genetische Information kann mittels Sequenzanalyse identifiziert werden. Quelle: „Workflow describing the ChECS screening“, P. Rathert, https://doi.org/10.1093/nar/gkab180, Oxford University Press, CC-BY-NC 4.0, bearbeitet von R. Menßen-Franz.Anschließend kombinierten die Forscher das Reportergen-System mit einem funktionellen genetischen Screen. Hierdurch lassen sich nicht nur direkte Interaktionspartner finden, sondern auch vorgeschaltete Proteine, die ebenfalls einen Einfluss auf die Aktivität des Enzyms haben können. In diesem Fall wurden die Zellen mit einer Vielzahl unterschiedlicher, kurzer sogenannter shRNA-Moleküle transduziert. Diese bilden kleine Haarnadelstrukturen (small hairpins) aus und interagieren mit den durch Gentranskription gebildeten mRNA-Molekülen der Zelle, sodass deren Translation in Proteine verhindert wird. Sind die so ausgeschalteten Proteine essenziell für die Funktion von LSD1, dann leuchten die Zellen auch nach Doxycyclin-Gabe weiterhin rot.
Neue regulatorische Kaskade identifiziert
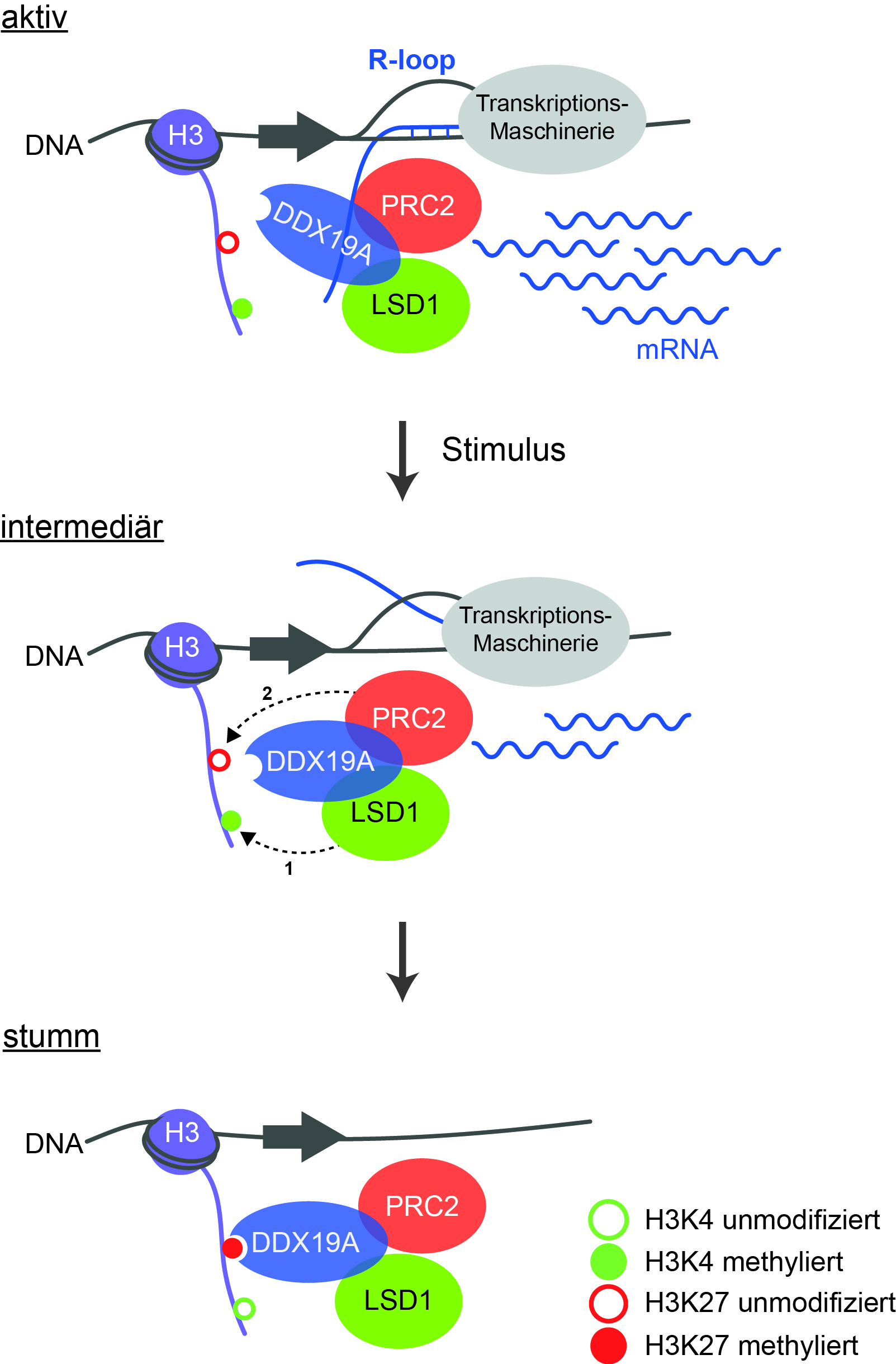 Modell der regulatorischen Kaskade zur Abschaltung von Genen. Während der aktiven Gentranskription ist H3K4 methyliert und es entstehen R-Loops. Nach einem externen Stimulus demethyliert LSD1 H3K4 (1), woraufhin PRC2 binden und K27 modifizieren kann (2). Dies kreiert eine Bindestelle für DDX19A, das die R-Loops vollständig entfernt.Quelle: “Model of the regulatory cascade for transcriptional repression“, P. Rathert, https://doi.org/10.1093/nar/gkab180, Oxford University Press, CC-BY-NC 4.0, bearbeitet von R. Menßen-Franz.
Modell der regulatorischen Kaskade zur Abschaltung von Genen. Während der aktiven Gentranskription ist H3K4 methyliert und es entstehen R-Loops. Nach einem externen Stimulus demethyliert LSD1 H3K4 (1), woraufhin PRC2 binden und K27 modifizieren kann (2). Dies kreiert eine Bindestelle für DDX19A, das die R-Loops vollständig entfernt.Quelle: “Model of the regulatory cascade for transcriptional repression“, P. Rathert, https://doi.org/10.1093/nar/gkab180, Oxford University Press, CC-BY-NC 4.0, bearbeitet von R. Menßen-Franz.Mit ihrer innovativen ChECS- (Chromatin Effector Coregulator Screen) Methode gelang es der Gruppe, neben bereits bekannten, auch neue Regulatoren von LSD1 zu identifizieren. Zu ihnen zählt DDX19A, eine Helikase, die spezielle Nukleinsäurestrukturen, sogenannte R-Loops, entwinden kann. Dies sind RNA-DNA-Hybride, die sich während der Transkription bilden und auch regulatorisch wirken. „Wir konnten erstmals den Zusammenhang zwischen epigenetischen Modifikationen und der Rolle von R-Loops bei der Inaktivierung von Genen zeigen“, berichtet der Biochemiker. Aktive Transkription geht mit einer Methylierung an Lysin 4 des Histon H3 (H3K4me) und der Bildung von R-Loops einher. Für das vollständige Ausschalten (Silencing) der Gene sind dann mehrere Schritte erforderlich: Nach Stimulation durch ein externes Signal entfernt LSD1 die Methylierung an K4. Nun kann der Polycomb Repressive Complex 2 (PRC2) binden und K27 methylieren, was einerseits bereits eine verringerte Transkription zur Folge hat und des Weiteren eine Bindungsstelle für die Helikase DDX19A erzeugt. Diese entfernt die noch verbleibenden R-Loops, was wiederum die Aktivität von LSD1 und PRC2 stimuliert. Ein robustes Silencing erfordert die Aktivität aller Komponenten der Kaskade.
Trotz unterschiedlicher initialer Mutationen besitzen viele Tumore einen ähnlichen Transkriptionsstatus, der unkontrollierte Zellteilung auslöst und von einer umprogrammierten epigenetischen Maschinerie aufrechterhalten wird. Aus diesem Grund geraten die regulatorischen epigenetischen Netzwerke immer mehr in den Fokus der Krebstherapie. Das von Rathert entwickelte neuartige Untersuchungssystem leistet einen entscheidenden Beitrag zur Entschlüsselung der komplexen Zusammenhänge und kann so eine Grundlage zur Entwicklung neuer Kombinationstherapien schaffen.