Automatisch Markiert
Epigenetische Schalter in Bakterien als Biosensoren
Die Analyse von Biomarkern für Krankheiten oder Pathogene kann für die Gesundheit entscheidend sein. Zum Nachweis bedarf es aber einer sensitiven und verlässlichen Methode, die schnelle Ergebnisse liefert. Solche Eigenschaften haben Biosensoren. Forscher am Institut für Biochemie und Technische Biochemie (IBTB) der Universität Stuttgart haben einen aus Plasmiden zusammengesetzten epigenetischen Schaltkreis konstruiert, der den Einsatz von Coli-Bakterien als Ganzzell-Biosensoren möglich machen könnte.
Die Epigenetik (griechisch „epi“ = jenseits) fasst Mechanismen zusammen, die einen DNA-Strang durch das Anknüpfen kleiner chemischer Moleküle verändern. Dadurch entsteht ein vererbbares epigenetisches Muster aus DNA-Modifikationen, das dazu beitragen, die Aktivität von Genen zu regulieren. Im Gegensatz zum genetischen Code, dessen Information in der Abfolge der DNA-Basen festgelegt und in jeder Körperzelle identisch ist, variieren epigenetische Muster je nach Zelltyp, können verändert werden und passen sich an unterschiedliche Umweltbedingungen an. Das Epigenom, also die Gesamtheit aller epigenetischen Modifikationen in einer Zelle, legt fest, welche Gene an- und abgeschaltet werden und somit, welche Funktionen die Zelle ausüben kann, ohne dass die DNA-Sequenz verändert wird.
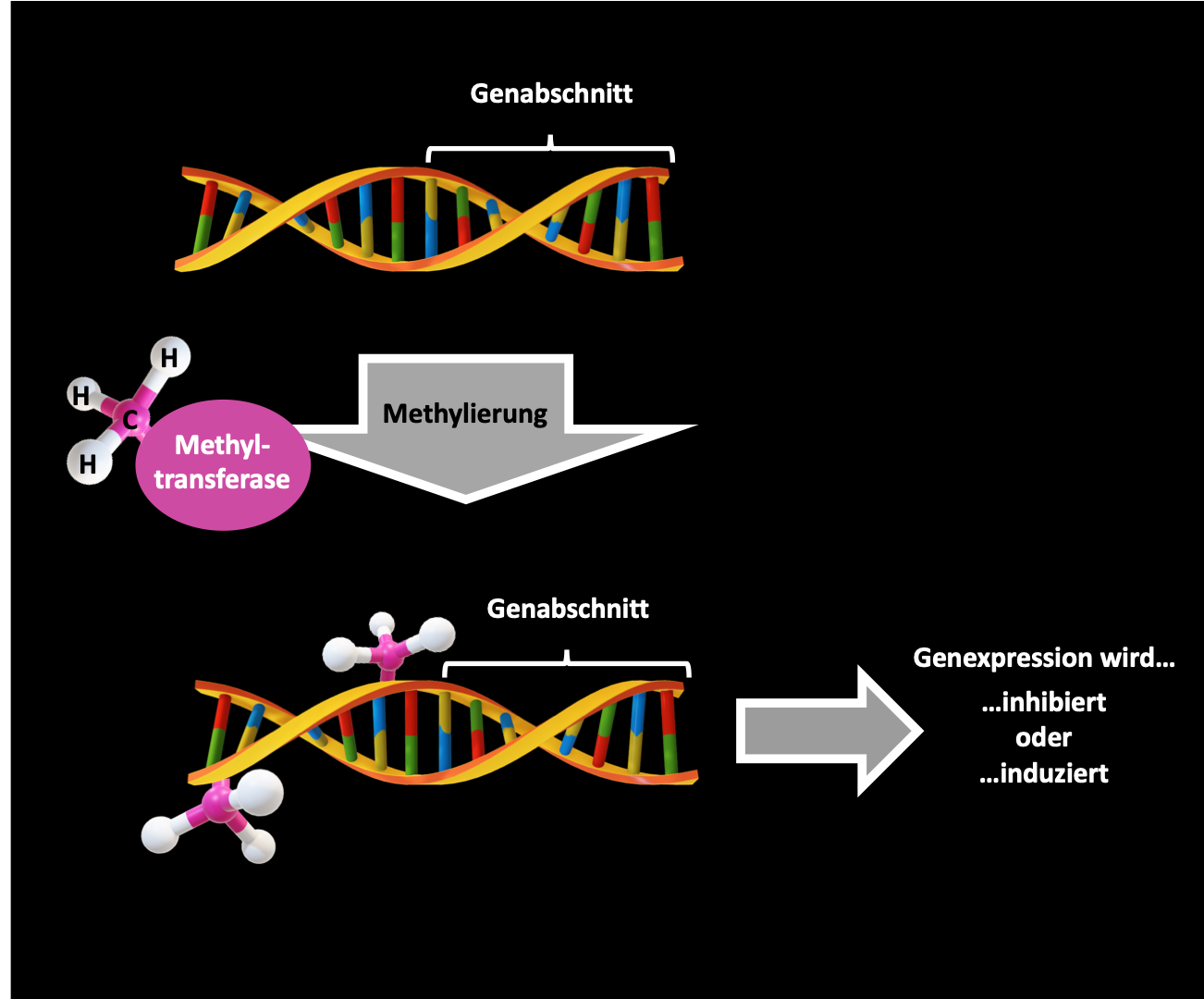 Genregulation über die Methylierung der DNA: Methyltransferasen können Methylgruppen (-CH3) an spezifische DNA-Basen anhängen und damit die Expression von Genen inhibieren oder induzieren. © BIOPRO Baden-Württemberg GmbH
Genregulation über die Methylierung der DNA: Methyltransferasen können Methylgruppen (-CH3) an spezifische DNA-Basen anhängen und damit die Expression von Genen inhibieren oder induzieren. © BIOPRO Baden-Württemberg GmbHDNA-Methylierung als epigenetische Markierung
Forscher unter der Leitung von Prof. Dr. Albert Jeltsch beschäftigen sich in der Abteilung Biochemie des Instituts für Biochemie und Technische Biochemie (IBTB) der Universität Stuttgart unter anderem mit der Erforschung und Optimierung von Enzymen, die an epigenetischen Prozessen beteiligt sind. Ein Beispiel für eine solche epigenetische Veränderung ist die DNA-Methylierung, wobei eine Methylgruppe (-CH3) enzymatisch durch sogenannte Methyltransferasen an eine spezifische Base in der DNA angeknüpft wird. Das Muster aus an die DNA angeknüpften Methylresten bewirkt in der Natur, dass manche Gene abgelesen, andere Gene stillgelegt werden.
Solche epigenetischen Prozesse finden nicht nur in Tieren, Pflanzen und Pilzen (Eukaryoten) statt, sondern auch in Bakterien (Prokaryoten), was sich die Stuttgarter Forscher um Jeltsch zunutze machen: Sie entwickeln künstliche Systeme in Escherichia coli -Bakterien, die diese befähigen, auf chemische und physikalische Signale aus ihrer Umwelt zu reagieren, indem sie diese Informationen in Form eines DNA-Methylierungsmusters speichern. Diese Bakterien könnten zukünftig als hoch sensitive Ganzzell-Biosensoren Anwendung finden, z. B. zur Detektion von Antibiotika im Trinkwasser und industriellen Schadstoffen in der Umwelt oder zur Analyse von Biomarkern für Krankheiten.
Antibiotika schon in geringsten Mengen aufspüren können
 Prof. Dr. Albert Jeltsch, Abteilungsleiter Biochemie und geschäftsführender Institutsleiter des IBTB an der Universität Stuttgart © Institut für Biochemie und Technische Biochemie (IBTB), Universität Stuttgart
Prof. Dr. Albert Jeltsch, Abteilungsleiter Biochemie und geschäftsführender Institutsleiter des IBTB an der Universität Stuttgart © Institut für Biochemie und Technische Biochemie (IBTB), Universität StuttgartEines der zukünftigen Anwendungsfelder, die Antibiotika, sind zwar für Mensch und Tier lebensrettende Medikamente, jedoch gelangen 30 bis 90 Prozent dieser Pharmazeutika über Urin und Kot in die Umwelt und damit letztlich auch wieder zurück in das Trinkwasser. So fördern sie auf lange Sicht die Entwicklung antibiotikaresistenter Pathogene, welche wiederum lebensbedrohlich sein können. Eine Anreicherung in der Umwelt muss deshalb so gut es geht verhindert werden. Allerdings mangelte es bis heute an sensitiven Nachweismöglichkeiten für die Arzneimittel, um Anreicherungsgebiete zu identifizieren und einschränkende Maßnahmen, wie beispielsweise den Einsatz spezieller Trinkwasserfilter, gezielt einleiten zu können.
Eine Möglichkeit, um zukünftig auch schon kleinste Mengen an Antibiotika kostengünstig aufzuspüren, könnten die künstlichen E.-coli-Systeme der Stuttgarter Wissenschaftler sein. Diese Bakterien, die ein Gedächtnissystem in sich tragen und zudem in eine geeignete Immobilisierungsvorrichtung eingebaut werden können, könnten so als Antibiotika-Ganzzell-Biosensoren minimale Antibiotikakontaminationen im Trinkwasser detektieren. Da Tetracyclin und seine Derivate in der Behandlung verschiedenster menschlicher Erkrankungen und darüber hinaus in der Tierhaltung Anwendung finden, wurde dieses Antibiotikum als zu detektierender Stoff gewählt. Wie die Forscher nachweisen konnten, besaß der Tetracyclin-Ganzzell-Biosensor eine äußerst sensitive Nachweisgrenze. „In unseren Tests konnten wir bereits 0,1 μg/l Tetracyclin aufspüren, was im Hinblick auf bereits in Kläranlagen gemessenen Tetracyclin-Restkonzentrationen von bis zu 2,37 μg/l interessant ist“, erklärt Institutsleiter Jeltsch. „Darüber hinaus war das System in der Lage, die einmalige Exposition mit Tetracyclin über mehrere Tage hinweg zu speichern.“ Diese Speicherfähigkeit ermöglicht Trinkwasseranalysen, die über mehrere Tage andauern können, wodurch die bei täglich auszuwechselnden Messinstrumenten anfallenden Kosten eingespart werden könnten.
Wie funktioniert der Bakterien-Biosensor?
Die biosensorischen Eigenschaften erhielten die Bakterien durch zwei verschiedene synthetisch hergestellte DNA-Plasmide, die ein Gedächtnissystem bilden. Die Aufnahme beider Plasmide erfolgte durch Elektroporation, eine Methode, die die bakterielle Membran über ein elektrisches Feld für kurze Zeit permeabel macht. In Anwesenheit des Antibiotikums Tetracyclin wird auf dem ersten, sogenannten Trigger-Plasmid zunächst die Produktion einer ersten DNA Methyltransferase eingeleitet. Diese methyliert eine bestimmte DNA-Sequenz auf dem Gedächtnis-Plasmid. Die Methylierung leitet wiederum die Bildung einer zweiten Methyltransferase in die Wege, wodurch die Anwesenheit von Tetracyclin auf dem Gedächtnis-Plasmid als Information gespeichert wird.
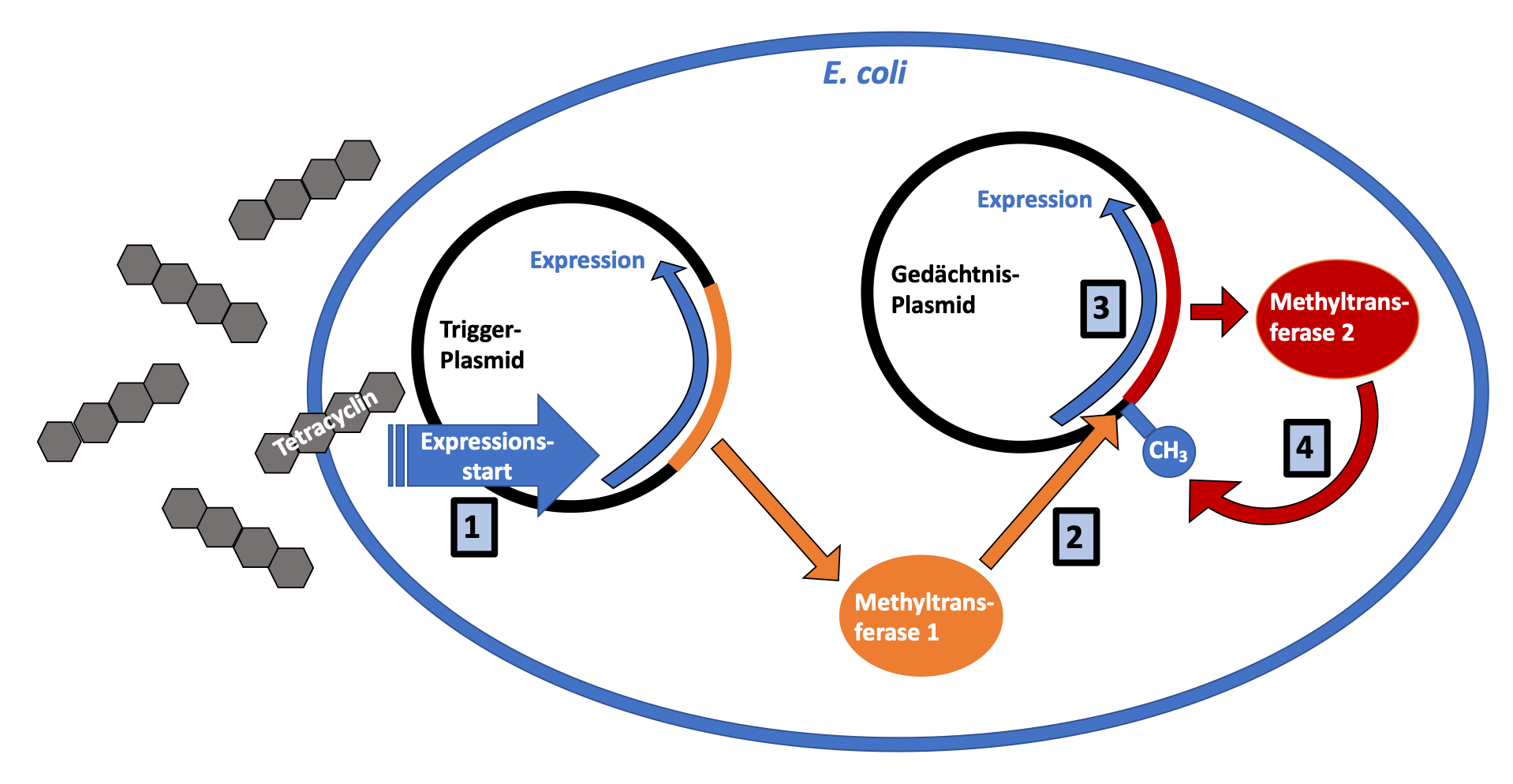 Das Tetracyclin-induzierbare epigenetische Gedächtnissystem in E. coli. 1) Tetracyclin führt zur Expressionsinduktion auf dem Trigger-Plasmid, woraufhin die erste Methyltransferase (Methyltransferase 1) produziert wird. 2) Methyltransferase 1 methyliert (blauer Lollipop mit Inschrift „CH3“: Methylgruppe(n)) das Gedächtnis-Plasmid. 3) In Folge der Methylierung durch Methyltransferase 1 startet die Expression der zweiten Methyltransferase (Methyltransferase 2). 4) Gedächtniseffekt: Die Methyltransferase 2 kann die Methylierung und damit die Expression des Gedächtnis-Plasmids aufrechterhalten (Maier et al., 2017). © BIOPRO Baden-Württemberg GmbH
Das Tetracyclin-induzierbare epigenetische Gedächtnissystem in E. coli. 1) Tetracyclin führt zur Expressionsinduktion auf dem Trigger-Plasmid, woraufhin die erste Methyltransferase (Methyltransferase 1) produziert wird. 2) Methyltransferase 1 methyliert (blauer Lollipop mit Inschrift „CH3“: Methylgruppe(n)) das Gedächtnis-Plasmid. 3) In Folge der Methylierung durch Methyltransferase 1 startet die Expression der zweiten Methyltransferase (Methyltransferase 2). 4) Gedächtniseffekt: Die Methyltransferase 2 kann die Methylierung und damit die Expression des Gedächtnis-Plasmids aufrechterhalten (Maier et al., 2017). © BIOPRO Baden-Württemberg GmbHDie Besonderheit dieses Systems liegt darin, dass das Expressionsverhalten der Methyltransferase einer positiven Rückkopplung entspricht. Denn sie kann ihre eigene Produktion nach dem einmaligen Kontakt des Gedächtnissystems mit Tetracyclin über mehrere Zellteilungen hinweg aufrechterhalten. Da der hierbei erzeugte epigenetische Gedächtniseffekt keinen Einfluss auf die genetische Information der Zelle nimmt, ist er im Hinblick auf die bisher entwickelten Gedächtnissysteme, die auf irreversiblen Veränderungen der DNA-Sequenz basieren, einzigartig. Darüber hinaus soll er von den Forschern in Zukunft noch weiter verbessert werden.
Mit der zuvor erwähnten positiven Rückkopplung geht die vorübergehend stetig ansteigende Konzentration des Enzyms einher. Somit reichen schon sehr geringe Mengen Tetracyclin, um ein starkes Signal des Systems hervorzurufen. Dementsprechend zeigt der Ganzzell-Biosensors eine hohe Sensitivität für Tetracyclin oder potenziell viele weitere Stoffe, wie zum Beispiel Krankheitsmarker im Körper.
Temperatur als Trigger
Das System umfasst außerdem eine temperatursensitive Komponente – ein Zinkfinger-Repressorprotein. Diese Proteine zeichnen sich durch ihre Zinkfingerdomäne aus, welche durch ein zentral gebundenes Zinkion eine schleifenförmige Struktur, den sogenannten Zinkfinger, annimmt und an der Regulation der Genexpression beteiligt ist. Mit dieser zusätzlichen temperatursensitiven Komponente könnte der Ganzzell-Biosensor neben der Anwendung als Umweltgift- und Krankheitsmarker-Detektor zudem zur Überprüfung von Kühlketten eingesetzt werden. Steigt die Temperatur während des Transports von Nahrungsmitteln oder Pharmazeutika im Lagerraum über einen Schwellenwert, wird der Schaltkreis aktiviert. Diese einmalige Überschreitung der Temperatur kann dank des Gedächtniseffekts des Systems auch noch nach mehreren Tagen nachgewiesen werden.
Das Zusammenspiel der hohen Empfindlichkeit, der unkomplizierten Umprogrammierbarkeit auf die Detektion verschiedenster chemischer und physikalischer Umweltreize und des einzigartigen Gedächtniseffektes macht den epigenetischen Ganzzell-Biosensor zu einem Werkzeug mit hohem Potenzial zur Ablösung bisheriger künstlicher Sensoren. Zur Umsetzung dieser Zukunftsvision möchten die Forscher in nächster Zeit an der Eliminierung falsch positiver Signale des Systems arbeiten und es damit noch stabiler konstruieren. Der finale Schritt nach noch nicht einschätzbaren Optimierungsarbeiten, wäre die Konstruktion einer geeigneten Immobilisierungseinrichtung, um das System außerhalb des Labors einsetzen zu können.
Epigenetik im Zusammenhang mit der Entstehung von Krankheiten
Auch in der Humanmedizin ist die Epigenetik ein hoch aktuelles Forschungsgebiet, so werden abnormale epigenetische Prozesse mit einer Vielzahl an Krebsarten und neurodegenerativen Erkrankungen in Verbindung gebracht. Beispielsweise wurde der Funktionsverlust oder die Funktionsänderungen von DNA Methyltransferasen, welche einen Teil des Epigenoms –das Methylierungsmuster der Zelle – aufrechterhalten, als karzinogene Faktoren identifiziert. Da das zellspezifische DNA Methylierungsmuster den Dichtegrad des Chromatins und damit einhergehend die Expressionsfrequenz bestimmt, führen fehlerhafte Methylierungen zur übermäßigen oder ausbleibenden Expression ganzer Genabschnitte – mit gravierenden Folgen wie der Entartung von Zellen oder der Zerstörung von Geweben. So besitzen spezifische Subtypen von Magen-, Darm- und Lungenkrebs charakteristische epigenetische Muster, welche als potenzielle Biomarker und überdies als Ansatzpunkte therapeutischer und präventiver Behandlungsarten in Frage kommen. Im Falle neurodegenerativer Erkrankungen, wie der Alzheimer- oder der Huntington-Krankheit sowie der Amyotrophen Lateralsklerose (ALS) konnten deutlich veränderte Transkriptionsprofile identifiziert werden, die auf eine fehlgeleitete Chromatinorganisation zurückzuführen waren.